近日,中国农业科学院农业基因组研究所农业微生物组学与营养健康创新团队开发了宏基因组分析新技术MetaKSSD,显著提升分析效率与准确性,助力发现更多微生物关联同时实现实时检测。相关研究成果发表在《自然·计算科学(Nature Computational Science)》上。
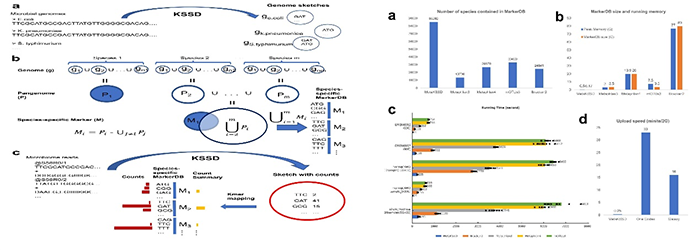
宏基因组分析依赖大规模标记数据库进行物种鉴定与丰度估计,但面临数据库规模膨胀、更新滞后及处理效率不足的挑战。传统方法依赖庞大的参考数据库,占用存储空间大、更新困难,且命令行、上传云端存在安全与带宽限制。随着基因组数据的爆炸式增长,亟需一种轻量化、用户友好的在线分析工具。
该研究开发了一种宏基因组分析新技术——MetaKSSD,该技术通过素描运算,可拓展性相比传统方法提升了53倍。在实际测试中,MetaKSSD还展示了显著的速度优势,以约11GB 的样本为例,完成其丰度分析仅需约11秒。为降低使用门槛,研究还提供MetaKSSD的客户端—服务器工作模式,用户可在有限带宽环境下实现实时在线分析。该研究对于高精度大规模宏基因组分析、宏基因组大样本数据汇集及宏基因组大模型构建具有重大意义。
该研究得到广东省实验室专项启动经费、广东省实验室科研任务多组学算法开发等项目的支持。(通讯员 马昕怡)
原文链接:
https://www.nature.com/articles/s43588-025-00855-0







